Bioinformática - PhyML: alinhamento de sequências nucleotídicas em ambiente paralelo
O PhyML é um software utilizado para se fazer estimativa e probabilidade filogênica em alinhamento de sequências nucleotídicas ou sequências de anino ácido. Atualmente está na versão 3.0, seu desenvolvimento teve participação de diversos cientistas de algumas universidades no mundo.
[ Hits: 31.602 ]
Por: José Cleydson Ferreira da Silva em 27/07/2010
Sumário - Sobre o PhyML
Sumário
1. Sobre o PhyML2. Menus do PhyML
2.1 Submenu de entrada de dados
2.2 Submenu de modelos de substituição
2.3 submenu - Pesquisa de Árvore
3. Usando PhyML em linha de comando
4. Instalando PHYML em ambiente paralelizado
4.1Download do PHYML
5.0 Sobre o autor
6.0 Referências
1. Sobre o PhyML
O PhyML é um software utilizado para se fazer estimativa e probabilidade filogênica em alinhamento de sequências nucleotídicas ou sequências de anino acido. Atualmente está na versão 3.0, seu desenvolvimento teve participação de diversos cientistas de algumas universidades como apresenta o quadro abaixo: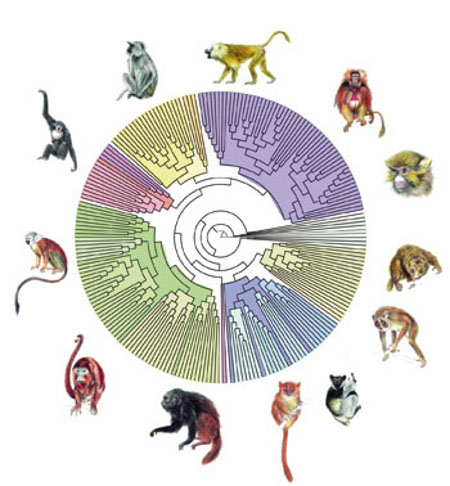
Departamento de Estatística. Universidade de Auckland. New Zealand
Universidade de Montpellier II - CNRS UMR 5506. LIRMM. Montpellier França.
e-mail : guindon@stat.auckland.ac.nz
Olivier Gascuel
Universidade Montpellier II - CNRS UMR 5506. LIRMM. Montpellier França.
e-mail : gascuel@lirmm.fr
Jean-Francois Dufayard
Universidade Montpellier II - CNRS UMR 5506. LIRMM. Montpellier França.
e-mail : jeanfrancois.dufayard@gmail.com
** Authors (before 2006)... **
Wim Hodrijk
Atualmente trabalhando na 'Rede nacional de Bioinformática - National Bioinformatics Network'. Cape Town, Africa do Sul
e-mail : wim@santafe.edu
Franck Lethiec
Universidade Montpellier II - CNRS UMR 5506. LIRMM. Montpellier França.
e-mail : lethiec@lirmm.fr
Este software é utilizado em linha de comando e possui uma interface simples com diversas diretivas de configuração e parâmetros que correspondem métodos estáticos, analise de sequência e entre outros.
Os métodos utilizado pelo PhyML dá-se também por Máxima Verosimilhança para reconstrução Filogenética, o que possibilita encontrar a árvore que mais provavelmente explicaria a evolução de determinado indivíduo . Em seguida podemos verificar os menus e suas funções.
2. Menus do PhyML
Os parâmetros de configuração podem ser utilizados de duas formas: Um por meio da interface do software e outro por meio de comandos shell. A interface do pode ser usada acessando menus conforme as letras que identificam as funções, a seguir podemos apresentá-las.2.1 Submenu de entrada de dados
[D] ............................... Data type (DNA/AA)Tipo de dados. Podendo ser DNA ou aminoácido.
[I] ...... Input sequences interleaved (or sequential)
Entrada de intercalada de sequência.
[M] ....................... Analyze multiple data sets
Analise múltipla de dados.
2.2 Submenu de modelos de substituição
[M] ................. Model of nucleotide substitutionModelo de substituição de Nucleotídios
[M] ................ Model of amino-acids substitution
Modelo de substituição de aminoácidos
[F] ................. Optimise equilibrium frequencies
Otimizar frequência
[F] . Amino acid frequencies (empirical/model defined)
Frequência de aminoácidos com modelo empírico definido
[T] .................... Ts/tv ratio (fixed/estimated)
Fixar ou estimar transcrição / transversão com relação a Máxima Verosimilhança
[V] . Proportion of invariable sites (fixed/estimated)
Proporção de viabilidade fixa ou estimada
[R] ....... One category of substitution rate (yes/no)
Categoria de substituição de taxa
[C] ........... Number of substitution rate categories
Numero de substituição da categoria taxas
2.3 submenu - Pesquisa de Árvore
[O] ........................... Optimise tree topologyOtimizar topologia da árvore
[S] .................. Tree topology search operations
Operações de pesquisa na topologia da árvore
[R] ......................... Use random starting tree
Iniciar árvore usando aleatoriedade
[N] .................. Number of random starting trees
Iniciar árvore com numero aleatório
[U] ..................... Input tree (BioNJ/user tree)
Entrar com uma árvore
2.4 Suporte a Brunch
[B] ................ Non parametric bootstrap analysisSem analise para métrica bootstrap
[A] ................ Approximate likelihood ratio test
Teste de aproximação Verosimilhança.
2. Usando PhyML em linha de comando
3. Instalando PHYML em ambiente paralelizado
4. Sobre autor
Bioinformática - Análise Filogenética com Clustalx
Conheça tudo sobre os hardwares que compõem o seu computador com um simples comando
Implementando servidor de aplicações PHP utilizando Zend Framework
Bing: Medindo velocidade da conexão no Linux
MetaTrader 5 facilita instalação em Linux
Instalando o Kink - Monitoração de tinta de impressoras HP e Epson
Instalando ou atualizando o seu KDE
aaPanel - Um Painel de Hospedagem Gratuito e Poderoso
Grande Kuruma!!
Parabéns pelo artigo. Apesar de eu não estar familiarizado com coisas como:
probabilidade filogênica
alinhamento de sequências nucleotídicas
sequências de anino ácido
Eu acho que isso irá e muito ajudar a comunidade do software livre.
E mais do que nunca
VIDA LONGA AO SL!!
Dailson Fernandes
http://www.dailson.com.br
Olá Dailson,
Grande Guerreiro, obrigado pelo incentivo meu camarada. Vou começar uma serie longa desses artigos, estou pensando em enviar um trabalho para o VOL DAY, em Bebedouro-SP, mas estou pensando se poderá ser aceito ou não. Esses artigos com certeza deram muito trabalho para validar a prática. Mas com certeza resolverá a vida dos Biologos e dos Bioinformatas.
Um abraço!
Patrocínio
Destaques
Artigos
Como atualizar sua versão estável do Debian
Cirurgia para acelerar o openSUSE em HD externo via USB
Void Server como Domain Control
Dicas
Script de montagem de chroot automatica
Atualizar Linux Mint 22.2 para 22.3 beta
Jogar games da Battle.net no Linux com Faugus Launcher
Como fazer a Instalação de aplicativos para acesso remoto ao Linux
Tópicos
Conky, alerta de temperatura alta (12)
De volta para o futuro - ou melhor, para o presente (parte 2) (2)
Por que passar nas disciplinas da faculdade é ruim e ser reprovado é b... (7)
Top 10 do mês
-

Xerxes
1° lugar - 148.617 pts -

Fábio Berbert de Paula
2° lugar - 67.175 pts -

Buckminster
3° lugar - 22.443 pts -

Mauricio Ferrari
4° lugar - 21.043 pts -
Alberto Federman Neto.
5° lugar - 20.075 pts -

edps
6° lugar - 18.961 pts -

Daniel Lara Souza
7° lugar - 18.014 pts -

Andre (pinduvoz)
8° lugar - 16.411 pts -

Alessandro de Oliveira Faria (A.K.A. CABELO)
9° lugar - 15.764 pts -

Jesuilton Montalvão
10° lugar - 14.967 pts




